Neue hochempfindliche qPCR-Kits zum Nachweis von Genmutationen
QClamp® detektiert seltene und umsetzbare Krebsmutationen und Fusionsgene mit höchster Sensitivität
Neue Technologie: Xenonukleinsäure (XNA) Molekulare Klammertechnologie
Das QClamp® Gene Mutation Detection Kit ist ein hochempfindlicher qPCR-Assay mit einer Nachweisempfindlichkeit von 0,1 % oder weniger. Er eignet sich ideal für das Screening von seltenen und verwertbaren somatischen Mutationen in Onkogenen. Der Assay verwendet eine sequenzspezifische Klammer (Xeno-Nukleinsäure-Sonde), die die PCR-Amplifikation der Wildtyp-DNA-Matrize unterdrückt und nur die mutierte Matrize selektiv amplifiziert. Die Kits stellen eine schnelle, reproduzierbare und erschwingliche Lösung dar, die einen einfachen Arbeitsablauf und PCR-Geräte verwendet, die üblicherweise in Forschungs- und klinischen Labors eingesetzt werden.

HOCHEMPFINDLICH
Nachweis von weniger als 0,1 % Mutationen
PROBENFORMAT
Geeignet für Flüssigbiopsie, FFPE-Gewebe und mehr
SCHNELL
Rationalisierter Arbeitsablauf mit 3 Stunden Durchlaufzeit
Gen
Mutationen
CE/IVD
Medizinprodukte für die In-vitro-Diagnostik. Lesen Sie die Gebrauchsanweisung sorgfältig durch.
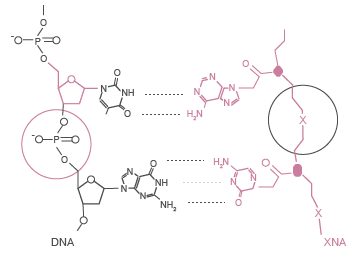
Xenonukleinsäure (XNA) Molekulare Klammertechnologie
DiaCarta-Wissenschaftler haben innovative neue molekulare Nukleinsäure-Oligomere entwickelt, die durch Watson-Crick-Basenpaarung an Ziel-DNA-Sequenzen hybridisieren, jedoch ein modifiziertes chemisches Grundgerüst besitzen. Die Xenonukleinsäure-Oligomere (Abbildung: Watson-Crick-Basenpaarung von DNA mit kognitiver XNA) hybridisieren hocheffektiv an Zielsequenzen und können als molekulare Klammern in quantitativen Echtzeit-Polymerase-Kettenreaktionen (PCR) oder als hochspezifische molekulare Sonden zum Nachweis von Nukleinsäure-Zielsequenzen eingesetzt werden.
MERKMALE UND VORTEILE
- Molekulare Klammern für qPCR
- Synthetische Oligomere mit natürlichem A, T, C, G oder modifizierten Nukleosiden (15 bis 25 nt lang)
- Hydrophiles und neutrales Grundgerüst (keine Phosphatgruppe wie PNA)
- Hybridisierung durch Watson-Crick-Paarung
- Resistent gegen alle bekannten Nukleasen
- Viel höhere Bindungsaffinität
- DNA-Bindung ist unabhängig von der Salzkonzentration
- Großer Schmelztemperaturunterschied (ΔTm=15-20ºC) bei Einzelnukleotiden (SNP's) und Insertionen/Deletionen (Indels) (5-7ºC für natürliche DNA)
Wie funktioniert es?
QClamp® ist eine revolutionäre neue Methode zum Screening auf somatische Mutationen, bei der eine sequenzspezifische Klammer verwendet wird, die die PCR-Amplifikation von Wildtyp-Template-DNA unterdrückt. QClamp® ermöglicht die selektive PCR-Amplifikation nur von mutierten Templates, was den Nachweis von mutierter DNA in Gegenwart eines großen Überschusses an Wildtyp-Templates aus einer Vielzahl von Proben ermöglicht, darunter FFPE, Flüssigbiopsie und traditionell schwierige Zytologieproben.
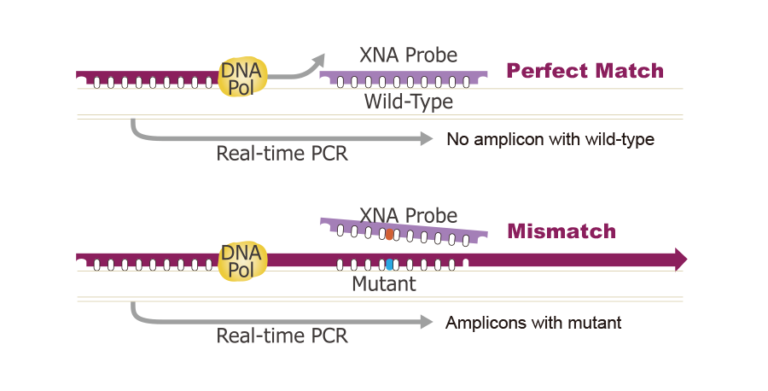
Unterstützende Daten
QClamp weist weniger als 0,1 % mutierte DNA nach
Die empfindlichste Technologie, die weniger als 0,1 % mutierte DNA in 2 Stunden nachweisen kann.
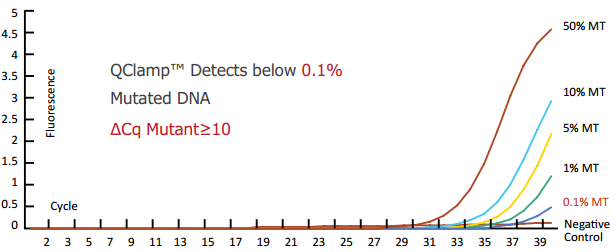
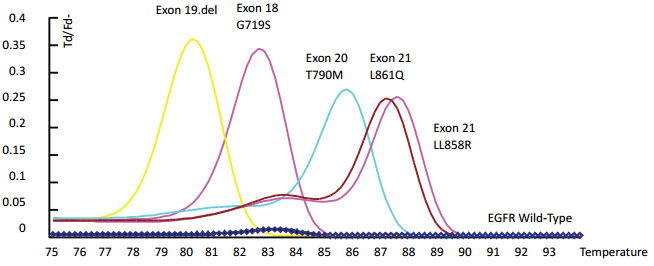
QClamp HRM für EGFR-Mutationen
Hochauflösende Schmelzprofile (HRM) von klinisch relevanten EGFR-Mutationen, die mit dem QClamp EGFR-Test erstellt wurden.